
- Автоматизация
- Антропология
- Археология
- Архитектура
- Биология
- Ботаника
- Бухгалтерия
- Военная наука
- Генетика
- География
- Геология
- Демография
- Деревообработка
- Журналистика
- Зоология
- Изобретательство
- Информатика
- Искусство
- История
- Кинематография
- Компьютеризация
- Косметика
- Кулинария
- Культура
- Лексикология
- Лингвистика
- Литература
- Логика
- Маркетинг
- Математика
- Материаловедение
- Медицина
- Менеджмент
- Металлургия
- Метрология
- Механика
- Музыка
- Науковедение
- Образование
- Охрана Труда
- Педагогика
- Полиграфия
- Политология
- Право
- Предпринимательство
- Приборостроение
- Программирование
- Производство
- Промышленность
- Психология
- Радиосвязь
- Религия
- Риторика
- Социология
- Спорт
- Стандартизация
- Статистика
- Строительство
- Технологии
- Торговля
- Транспорт
- Фармакология
- Физика
- Физиология
- Философия
- Финансы
- Химия
- Хозяйство
- Черчение
- Экология
- Экономика
- Электроника
- Электротехника
- Энергетика
Манипуляции с таблицами и массивами.
Манипуляции с таблицами и массивами.
sort() – сортировка вектора,
rank() – определение рангов элементов вектора,
order() – определение порядковых номеров элементов вектора,
Индексы, выборка строк и столбцов. Выборка произвольной последовательности элементов. Логические индексы для матриц. Использование индексов в левой части присваивания.
match() – поиск соответствия
merge() – слияние таблиц
Предположения об исходных данных:
нормальность:
qqplot(),
qqnorm(),
hist()+plot(dnorm()),
однородность дисперсии:
boxplot()
var. test()
var. test(x, y, ratio = 1,
alternative = c(" two. sided", " less", " greater" ),
conf. level = 0. 95, ... )
Модели:
линейные (регрессия):
нелинейные (аддитивные):
формула:
x1+x2, x1*x2, g/x, g*x, g: x
модельная матрица:
model. matrix(~x1+x2)
факторы и контрасты.
pairs()
оценка параметров модели:
summary()
Описание полей вывода
Call:
lm(formula = abundance ~ NAP * fBeach, data = p1)
Residuals:
Min 1Q Median 3Q Max
-222. 41 -12. 83 -1. 24 11. 78 395. 37
Coefficients:
Estimate Std. Error t value Pr(> |t|)
(Intercept) 141. 77 60. 33 2. 350 0. 0263 *
NAP 74. 27 70. 06 1. 060 0. 2985
fBeach2 119. 07 79. 97 1. 489 0. 1481
fBeach3 -115. 57 78. 44 -1. 473 0. 1522
fBeach4 -94. 78 83. 84 -1. 130 0. 2682
fBeach5 -104. 31 88. 11 -1. 184 0. 2468
fBeach6 -106. 12 79. 11 -1. 341 0. 1910
fBeach7 57. 23 102. 25 0. 560 0. 5803
fBeach8 -87. 56 81. 31 -1. 077 0. 2911
fBeach9 -118. 61 84. 25 -1. 408 0. 1706
NAP: fBeach2 167. 57 90. 17 1. 858 0. 0741.
NAP: fBeach3 -91. 14 92. 27 -0. 988 0. 3320
NAP: fBeach4 -95. 32 88. 31 -1. 079 0. 2899
NAP: fBeach5 -96. 54 96. 50 -1. 000 0. 3260
NAP: fBeach6 -68. 51 82. 71 -0. 828 0. 4148
NAP: fBeach7 -196. 54 102. 90 -1. 910 0. 0668.
NAP: fBeach8 -95. 78 82. 00 -1. 168 0. 2530
NAP: fBeach9 -84. 36 88. 35 -0. 955 0. 3481
---
Signif. codes: 0 ‘***’ 0. 001 ‘**’ 0. 01 ‘*’ 0. 05 ‘. ’ 0. 1 ‘ ’ 1
Residual standard error: 112. 1 on 27 degrees of freedom
Multiple R-squared: 0. 6634, Adjusted R-squared: 0. 4515
F-statistic: 3. 13 on 17 and 27 DF, p-value: 0. 003985
coef()
fitted()
residuals()
predict()
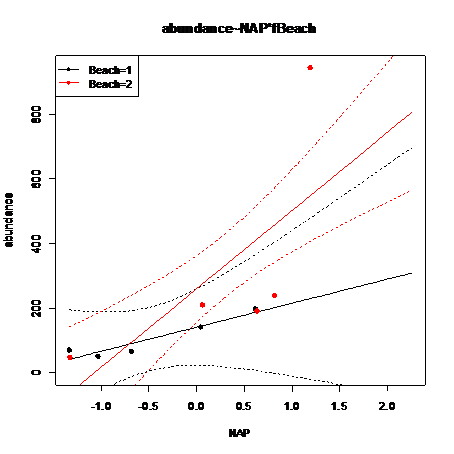
tmp. lim< -range(p1$NAP)
tmp. NAP< -seq(tmp. lim[1], tmp. lim[2], len=100)
tmp. df < -data. frame(NAP=tmp. NAP, fBeach=factor(1, levels=levels(p1$fBeach)))
tmp. aban1< -predict(p. lm4, newdata=tmp. df, se. fit=TRUE)
plot (tmp. NAP, tmp. aban1$fit, main=" abundance~NAP*fBeach", xlab=" NAP", ylab=" abundance", lwd=2, type=" l", ylim=c(0, max(p1$abundance)))
points(tmp. NAP, tmp. aban1$fit+1. 96*tmp. aban1$se. fit, lty=2, type=" l" )
points(tmp. NAP, tmp. aban1$fit-1. 96*tmp. aban1$se. fit, lty=2, type=" l" )
ind< -p1$fBeach==1
points(p1$NAP[ind], p1$abundance[ind], pch=20, cex=1. 5)
tmp. df < -data. frame(NAP=tmp. NAP, fBeach=factor(2, levels=levels(p1$fBeach)))
tmp. aban1< -predict(p. lm4, newdata=tmp. df, se. fit=TRUE)
points(tmp. NAP, tmp. aban1$fit, lwd=2, type=" l", col=" red" )
points(tmp. NAP, tmp. aban1$fit+1. 96*tmp. aban1$se. fit, lty=2, type=" l", col=" red" )
points(tmp. NAP, tmp. aban1$fit-1. 96*tmp. aban1$se. fit, lty=2, type=" l", col=" red" )
ind< -p1$fBeach==2
points(p1$NAP[ind], p1$abundance[ind], pch=20, cex=1. 5, col=" red" )
legend(" topleft", c(" Beach=1", " Beach=2" ), pch=20, lwd=2, col=c(" black", " red" ))
|
|
|
|
© helpiks.su При использовании или копировании материалов прямая ссылка на сайт обязательна.
|