
- Автоматизация
- Антропология
- Археология
- Архитектура
- Биология
- Ботаника
- Бухгалтерия
- Военная наука
- Генетика
- География
- Геология
- Демография
- Деревообработка
- Журналистика
- Зоология
- Изобретательство
- Информатика
- Искусство
- История
- Кинематография
- Компьютеризация
- Косметика
- Кулинария
- Культура
- Лексикология
- Лингвистика
- Литература
- Логика
- Маркетинг
- Математика
- Материаловедение
- Медицина
- Менеджмент
- Металлургия
- Метрология
- Механика
- Музыка
- Науковедение
- Образование
- Охрана Труда
- Педагогика
- Полиграфия
- Политология
- Право
- Предпринимательство
- Приборостроение
- Программирование
- Производство
- Промышленность
- Психология
- Радиосвязь
- Религия
- Риторика
- Социология
- Спорт
- Стандартизация
- Статистика
- Строительство
- Технологии
- Торговля
- Транспорт
- Фармакология
- Физика
- Физиология
- Философия
- Финансы
- Химия
- Хозяйство
- Черчение
- Экология
- Экономика
- Электроника
- Электротехника
- Энергетика
Matériels et méthodes
Maté riels et mé thodes
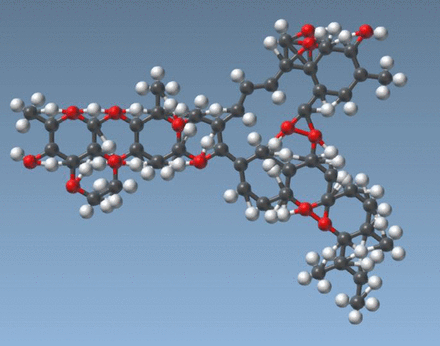
Nous avons utilisé le programme AutoDock Vina Extended pour effectuer l'é tude d'amarrage ( 7 ). La molé cule d'ivermectine provient de PubChem CID: 6321424 ( Figure 1 ). La structure cristalline du domaine de liaison au ré cepteur de pointe SARS-CoV-2 lié à l'ACE2 a é té dé posé e dans la banque de donné es sur les proté ines 2020-02-21, publié e le: 2020-03-18 ( 8 ). Nous avons analysé le ré cepteur humain ACE2 car cette enzyme diffè re apparemment selon les espè ces, et l'affinité du virus pour la forme humaine peut expliquer son infectivité particuliè re pour l'homme ( 9 ).
- Afficher en ligne
- Afficher la fenê tre contextuelle
- Té lé charger powerpoint
Tableau I.
Paramè tres d'amarrage calculé s par AutoDock Vina Extended.
Ré sultats
Les é carts par rapport à la racine carré e moyenne des positions atomiques (DSMR en Angstroms) sont indiqué es dans le tableau I. Des valeurs plus faibles de RMSD indiquent que l'amarrage est validé avec une plus grande pré cision. Les valeurs RMSD de 3 ou plus indiquent qu'aucun amarrage n'a eu lieu. Une seule position d'amarrage avec RMSD=0 est hautement valide, et cette position d'amarrage est illustré e sur les figures 2 et 3. Les 9 calculs d'amarrage suivants é taient supé rieurs à 22 Angstroms RMSD, indiquant qu'aucun amarrage n'a é té dé tecté ( Figure 4 et Tableau I ).
L'ivermectine s'est amarré e dans la ré gion de la leucine 91 du pic et de l'histidine 378 du complexe ré cepteur SARS Cov2-ACE2, entre la proté ine SARS-Cov2 et la proté ine ACE2. L'é nergie de liaison de l'ivermectine au complexe pointe-ACE2 é tait de -18 kcal/mol et la constante de liaison é tait de 5, 8 × 10 -8.
Des mé thodes biochimiques computationnelles et des logiciels d'amarrage ont é té utilisé s pour cribler des mé dicaments potentiels dans les sites de proté ines structurelles et de proté ines non structurelles du SRAS-Cov2. La ribavirine (un mé dicament antiviral courant), le remdesivir, la chloroquine et la luté oline ont é té analysé s ( 10 ). Les principaux inhibiteurs de la proté ase du SRAS-CoV-2 ont é té pré dits à l'aide d'Autodock ( 11 ), parmi lesquels l'hispidine et la lé pidine E, deux composé s naturels, et l'acide folique ( 12 ).
- Té lé charger la figure
- Ouvrir dans un nouvel onglet
- Té lé charger powerpoint
|
|
|
© helpiks.su При использовании или копировании материалов прямая ссылка на сайт обязательна.
|