
- Автоматизация
- Антропология
- Археология
- Архитектура
- Биология
- Ботаника
- Бухгалтерия
- Военная наука
- Генетика
- География
- Геология
- Демография
- Деревообработка
- Журналистика
- Зоология
- Изобретательство
- Информатика
- Искусство
- История
- Кинематография
- Компьютеризация
- Косметика
- Кулинария
- Культура
- Лексикология
- Лингвистика
- Литература
- Логика
- Маркетинг
- Математика
- Материаловедение
- Медицина
- Менеджмент
- Металлургия
- Метрология
- Механика
- Музыка
- Науковедение
- Образование
- Охрана Труда
- Педагогика
- Полиграфия
- Политология
- Право
- Предпринимательство
- Приборостроение
- Программирование
- Производство
- Промышленность
- Психология
- Радиосвязь
- Религия
- Риторика
- Социология
- Спорт
- Стандартизация
- Статистика
- Строительство
- Технологии
- Торговля
- Транспорт
- Фармакология
- Физика
- Физиология
- Философия
- Финансы
- Химия
- Хозяйство
- Черчение
- Экология
- Экономика
- Электроника
- Электротехника
- Энергетика
Задание 1: построение изображения структуры кристалла, проведение стандартных геометрических расчетов, анализ системы химических связей.
Модуль 3. Работа с программой IsoCryst. Визуализиция кристаллических структур. Геометрический и топологический анализы.
Задание 1: построение изображения структуры кристалла, проведение стандартных геометрических расчетов, анализ системы химических связей.
Инструменты: программы AutoCN и IsoCryst, карточка Crystal Data.
Пример 1: анализ кристаллической структуры b-UO2SO4.
Алгоритм:
(1) Откройте базу данных UO2SO4, которая содержит одну запись, отвечающую b-UO2SO4 и рассчитайте матрицу смежности при помощи программы AutoCN (Method: Distances).
(3) Откройте окно программы IsoCryst (Programs/IsoCryst). Или нажмите на кнопку 
(4) Выполните следующие действия с изображением кристаллической структуры:
- Нажмите кнопку
 , чтобы нарисовать содержимое элементарной ячейки.
, чтобы нарисовать содержимое элементарной ячейки. - «Выращивайте» структуру (то есть добавьте к содержимому элементарной ячейки атомы, связанные с ними за пределами ячейки). Используйте команду Ctrl+W или нажмите кнопку
 один раз. Изображение будет похоже на приведенное ниже.
один раз. Изображение будет похоже на приведенное ниже.
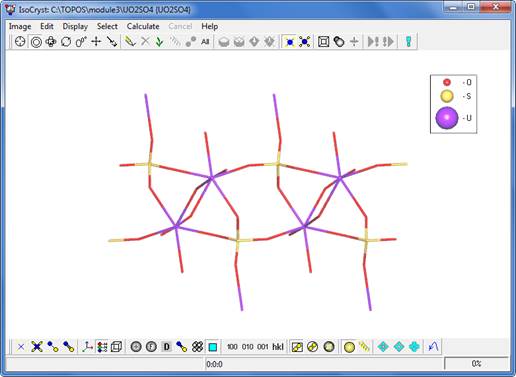
- Нажмите на кнопку
 или на кнопку
или на кнопку  для выбора режима изображения OpenGL или API, соответственно.
для выбора режима изображения OpenGL или API, соответственно. - Посмотрите различные модели изображения атомов и связей: Тонкие линии – Цилиндрические линии – Сферы и Тонкие линии – Сферы и Цилиндрические линии
 .
. - Попробуйте получить окрашенные связи в отличие от неокрашенных (белых)
 .
.
- Выполните переключения включено/выключено: Показать кристаллографические оси – Показать легенду – Показать ребра элементарной ячейки
 .
. - Осуществите перемещение изображения с помощью инструментов Вращение – Вращение в плоскости рисунка – Увеличение/Уменьшение – Перемещение в плоскости рисунка
 . Для вращения или увеличения на определенную величину, дважды щелкните на иконке инструмента и введите нужную величину.
. Для вращения или увеличения на определенную величину, дважды щелкните на иконке инструмента и введите нужную величину. - Некоторые из оставшихся в нижней части кнопок предусматривают отображение структуры согласно определенным заданным направлениям/проекциям, характеризующим кристаллографические направления hkl:
 ,
,  или
или  (по оси a, b или c). Нажав на кнопку
(по оси a, b или c). Нажав на кнопку  , Вы можете задать значения hkl для получения проекции в нужном направлении, например, 1,0,1 или 0,-1,3 и т.д.
, Вы можете задать значения hkl для получения проекции в нужном направлении, например, 1,0,1 или 0,-1,3 и т.д. - Выведите на рисунок имена атомов, нажав кнопку
 .
. - Нажмите на кнопку
 для использования инструмента Select. Выделите один из атомов урана и оставьте на рисунке только его, нажав кнопку
для использования инструмента Select. Выделите один из атомов урана и оставьте на рисунке только его, нажав кнопку  .
. - Выполните один раз команду рост структуры нажатием кнопки
 и переместите изображение в центр, нажав клавишу Space (пробел) на клавиатуре.
и переместите изображение в центр, нажав клавишу Space (пробел) на клавиатуре. - Изобразите координационный полиэдр атома урана (он должен быть выделен), нажав кнопку
 . Удалите изображение полиэдра с помощью кнопки
. Удалите изображение полиэдра с помощью кнопки  .
.
Рассчитайте межатомные расстояния U–O. Для этого, выделив атом урана, выделяйте атомы кислорода один за другим, каждый раз снимайте выделение с предыдущего атома кислорода. Значение межатомного расстояния Вы увидите в статус-строке. Например, r (O1-U1) = 2.361 Å, r (O3-U1) = 2.32 Å.
· Рассчитайте валентные углы O–U–O, выделяя тройки атомов. Каковы значения углов для экваториальных и аксиальных атомов кислорода? Например, угол (O5-U1-O6) = 176.11° для аксиальных и 83.76° для экваториальных атомов.
- Снимите выделение со всех атомов, щелкнув правой кнопкой мыши в свободном месте окна.
- Рассчитайте среднеквадратичное отклонение (s) от МНК-плоскости для экваториальных атомов кислорода координационной сферы атома урана, включая атом урана. Для этого выделите все эти атомы и выполните команду Calculate/Selected Atoms. В результате получим значение s = 0.15Å. Рассчитайте расстояние от атома урана до МНК-плоскости, выделив его малиновым цветом (с нажатой клавишей Ctrl). Вы можете выделить атомы кислорода экваториальной плоскости желтым цветом (щелчок на атоме левой кнопкой мыши) и атом урана – малиновым (щелчок на атоме левой кнопкой мыши с нажатой клавишей Ctrl). Результат в статус-строке должен быть равен 0.0492Å. Снимите выделение со всех атомов, щелкнув правой кнопкой мыши в свободном месте рабочего окна.
- Рассчитайте угол между экваториальной плоскостью координационного полиэдра атома урана и аксиальной группой O6–U1–O6. Вы должны выделить атомы кислорода экваториальной плоскости и уранильную группу желтым и малиновым цветом, соответственно. Угол будет равен 88.80°.
- Восстановите первоначальное изображение, нажав кнопку
 .
. - Выполните один раз команду Growth(Рост). Выделите все атомы, содержащиеся в сфере радиуса 3Å вокруг одного из атомов урана. Для этого установите режим выделения в сфере (
 ), выделите атом урана и при нажатой левой кнопки мыши протаскивайте курсор мыши пока не получите сферу требуемого радиуса. Вы получите 8 выделенных атомов. Нажмите кнопку
), выделите атом урана и при нажатой левой кнопки мыши протаскивайте курсор мыши пока не получите сферу требуемого радиуса. Вы получите 8 выделенных атомов. Нажмите кнопку  , чтобы оставить на рисунке только выделенные атомы.
, чтобы оставить на рисунке только выделенные атомы.
Выполните один раз командуGrowthи рассчитайте торсионные углыO–U–O–S. Найдите угол равный 148.08°. Ответ: O3-U1-O2-S1.
- Рассчитайте угол между экваториальными плоскостями соседних атомов урана, связанных через мостиковый атом кислорода. Для этого выделите атомы кислорода этих двух плоскостей желтым и малиновым цветом. Угол будет равен 61.16°.
- Восстановите исходное изображение структуры нажатием кнопки
 и выполните командуGrowthнесколько раз.
и выполните командуGrowthнесколько раз. - Выделите цепочки координационных полиэдров атомов урана, связанных вершинами (атомы кислорода). Для этого выведите на экран легенду (список атомов), нажав кнопку
 , затем выделите малиновым цветом все атомы серы, щелкнув на атоме серы в легенде и удерживая нажатую клавишу Ctrl. Удалите выделенные атомы серы нажатием клавиши Delete. Затем выделите все атомы в одной цепочке, перейдя в режим выделения
, затем выделите малиновым цветом все атомы серы, щелкнув на атоме серы в легенде и удерживая нажатую клавишу Ctrl. Удалите выделенные атомы серы нажатием клавиши Delete. Затем выделите все атомы в одной цепочке, перейдя в режим выделения  и щелкнув на любом атоме в цепи.
и щелкнув на любом атоме в цепи. - Восстановите исходное изображение структуры нажатием кнопки
 и выполните командуGrowthнесколько раз.
и выполните командуGrowthнесколько раз. - Выделите в структуре псевдослои, удалив атомы O(2). Для этого выберите инструмент “Magic Wand” (“волшебная палочка”)
 , щелкните на одном из атомов O(2) и установите Command: Delete, Do for: Cryst. Sort. Выделите один слой и оставьте на рисунке только его. Ориентируйте этот слой перпендикулярно направлению [001], используя кнопку
, щелкните на одном из атомов O(2) и установите Command: Delete, Do for: Cryst. Sort. Выделите один слой и оставьте на рисунке только его. Ориентируйте этот слой перпендикулярно направлению [001], используя кнопку  . Восстановите исходное изображение структуры.
. Восстановите исходное изображение структуры.
Выделите в структуре другие псевдослои, удалив атомы O(3). Какому направлению они перпендикулярны? Ответ: [100].
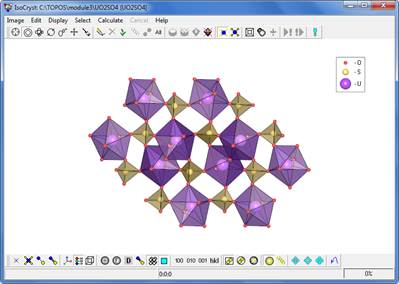
- Восстановите исходное изображение структуры и выполните командуGrowthнесколько раз. Выделите все атомы урана и серы и изобразите структуру в полиэдрическом представлении, используя кнопку
 . Рассмотрите различные варианты изображения полиэдров в окне опций IsoCryst (используйте кнопку
. Рассмотрите различные варианты изображения полиэдров в окне опций IsoCryst (используйте кнопку  , раздел Polyhedra).
, раздел Polyhedra).
- Удалите изображение полиэдров, выделите все атомы урана и удалите их. Используйте команду Undo (кнопка
 ) для отмены удаления.
) для отмены удаления. - Закройте окно программы IsoCryst.
Пример 2: анализ полиморфной модификации гидрохинона.
Алгоритм:
(1) Откройте базу данных C6H6O2 и вычислите матрицы смежности для всех 11 соединений состава C6H6O2, учитывая водородные связи (AutoCN Method: Domains).
(2) Перейдите ко второй записи в списке (Ref. Code HYQUIN), откройте карточку Crystal Dataиубедитесь, что это полиморфная модификация гидрохинона.
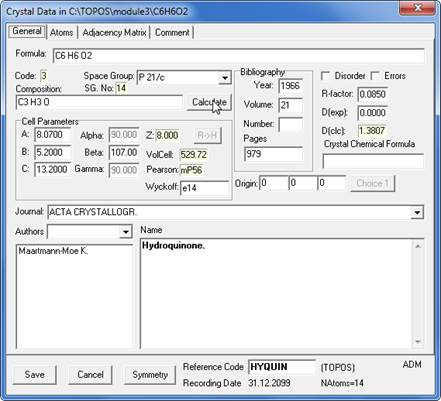
Нажмите кнопку Calculateи установить, что состав элементарной ячейки соответствует составу гидрохинона. Нажмите кнопку Yes.
(3) Откройте окно программы IsoCryst (Programs/IsoCryst). Выполните следующие операции:
- Выведите на экран изображение одной элементарной ячейки, нажав на кнопку желтый карандаш
 .
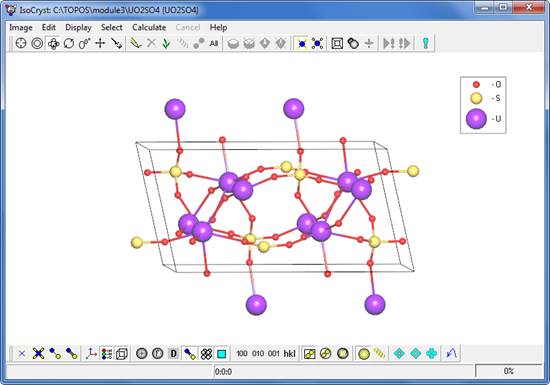
. - «Выращивайте» структуру (то есть добавьте к содержимому элементарной ячейки атомы, связанные с ними за пределами ячейки). Используйте команду Ctrl+W или нажмите кнопку
 . Изображение будет похоже на приведенное ниже.
. Изображение будет похоже на приведенное ниже.
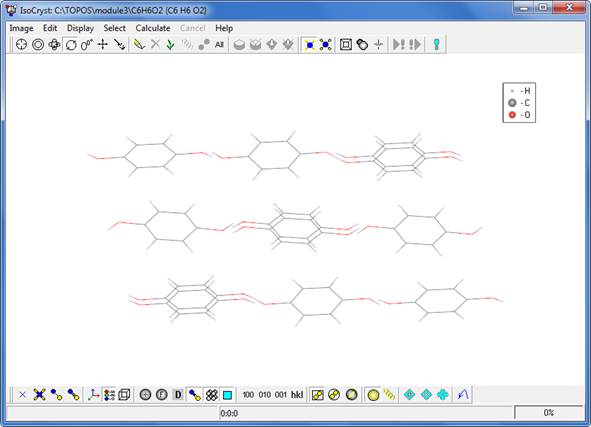
- Нажмите на кнопку
 или на кнопку
или на кнопку  для выбора режима изображения OpenGL или API, соответственно.
для выбора режима изображения OpenGL или API, соответственно. - Посмотрите различные модели изображения атомов и связей: Тонкие линии – Цилиндрические линии – Сферы и Тонкие линии – Сферы и Цилиндрические линии
 .
. - Выполните переключения включено/выключено: Показать кристаллографические оси – Показать легенду – Показать ребра элементарной ячейки
 .
. - Осуществите перемещение изображения с помощью инструментов Вращение – Вращение в плоскости рисунка – Увеличение/Уменьшение – Перемещение в плоскости рисунка
 .
. - Некоторые из оставшихся в нижней части кнопок предусматривают отображение структуры согласно определенным заданным направлениям/проекциям, характеризующим кристаллографические направления hkl:
 ,
,  или
или  (по оси a, b или c). Нажав кнопку
(по оси a, b или c). Нажав кнопку  , Вы можете задать значения hkl для получения проекции в нужном направлении, например, 1,0,1 или 0,-1,3 и т.д..
, Вы можете задать значения hkl для получения проекции в нужном направлении, например, 1,0,1 или 0,-1,3 и т.д..
Нажмите на кнопку  для использования инструмента Select. Выделите одну группу атомов, связанных друг с другом. Для этой цели активизируйте кнопку
для использования инструмента Select. Выделите одну группу атомов, связанных друг с другом. Для этой цели активизируйте кнопку  “select polymeric chain” (“выделить полимерную цепь”) и затем щелкните на атоме, все остальные атомы, связанные с ним будут выделены и окрашены в желтый цвет. Это сразу покажет, что соединение состоит из 0D, 1D или 3D подсеток.
“select polymeric chain” (“выделить полимерную цепь”) и затем щелкните на атоме, все остальные атомы, связанные с ним будут выделены и окрашены в желтый цвет. Это сразу покажет, что соединение состоит из 0D, 1D или 3D подсеток.
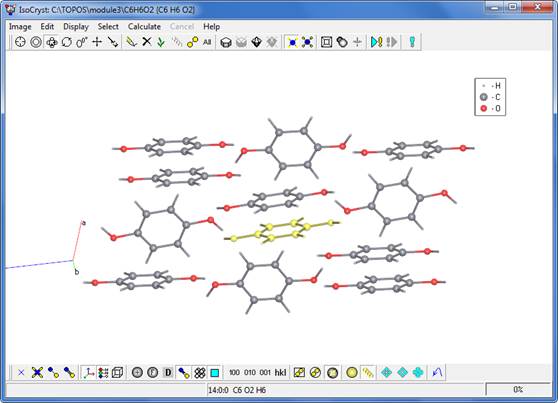
- Выберите изображение только выделенной подсетки, нажав клавишу
 (Show selection only- Показывать только выделенное). Выведите на экран одновременно и имена атомов и величины межатомных контактов нажатием некоторых кнопок, расположенных внизу слева:
(Show selection only- Показывать только выделенное). Выведите на экран одновременно и имена атомов и величины межатомных контактов нажатием некоторых кнопок, расположенных внизу слева:  (Atoms labels - Имена атомов) и
(Atoms labels - Имена атомов) и  (Show Bond Distances – Показывать длины связей). Нажмите на кнопку
(Show Bond Distances – Показывать длины связей). Нажмите на кнопку  , чтобы показать невыделенные атомы.
, чтобы показать невыделенные атомы. -
Нарисуйте другие типы связей/взаимодействий. Вы должны зайти в карточку Optionsследующим образом Image/Options или нажмите справа кнопку . Зайдите в раздел Bonds и выделите те типы связи, которые хотите сделать видимыми, например, водородные связи (колонка Show). Нажмите Ok.
. Зайдите в раздел Bonds и выделите те типы связи, которые хотите сделать видимыми, например, водородные связи (колонка Show). Нажмите Ok.
Водородные связи отмечены пунктирной линией.
- Нажмите кнопку
 и отключите
и отключите  и
и  . Затем снова откройте карточку Options. Установите флаг Hydrogen bondsв колонке Take для учета водородных связей по мере роста структуры.
. Затем снова откройте карточку Options. Установите флаг Hydrogen bondsв колонке Take для учета водородных связей по мере роста структуры.
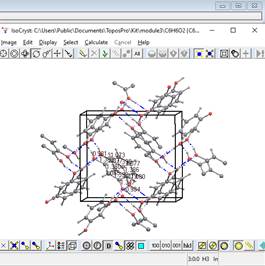
Нажмите Ok и выполните один раз команду Growth, чтобы получить атомы, связанные с выделенной молекулой водородными связями.
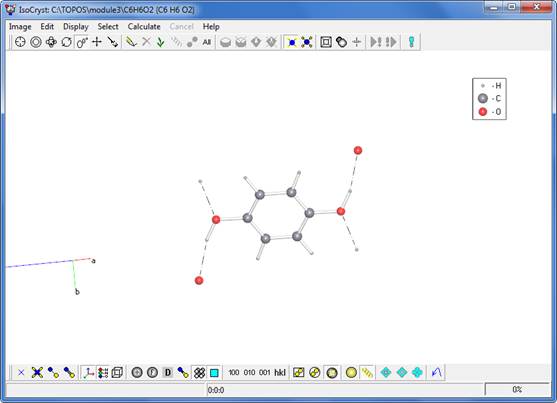
Теперь очистите флаг Hydrogen bonds, снимите выделение с молекулы, щелкнув правой кнопкой мыши на любом из ее атомов или в свободном месте рабочего окна и выполните команду Growth несколько раз, чтобы получить молекулы, связанные с выделенной молекулой водородными связями.
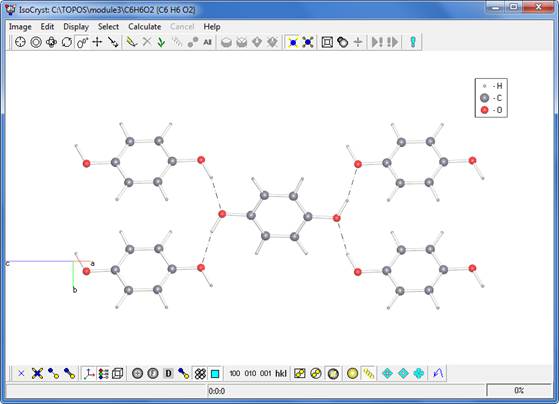
Снова установите флаг Hydrogen bonds и выполните команду роста сетки. Теперь нажмите клавишу Space (пробел) или кнопку  , чтобы вписать изображение в размер окна. Повращайте изображение и убедитесь, что получилась 2D двумерная сетка.
, чтобы вписать изображение в размер окна. Повращайте изображение и убедитесь, что получилась 2D двумерная сетка.
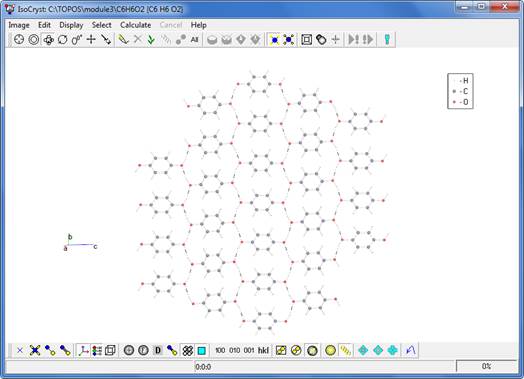
Нажмите кнопку Undo  несколько раз, чтобы вернуться к предыдущему изображению.
несколько раз, чтобы вернуться к предыдущему изображению.
- Для вычисления расстояний, углов или торсионных углов включите режим выбора
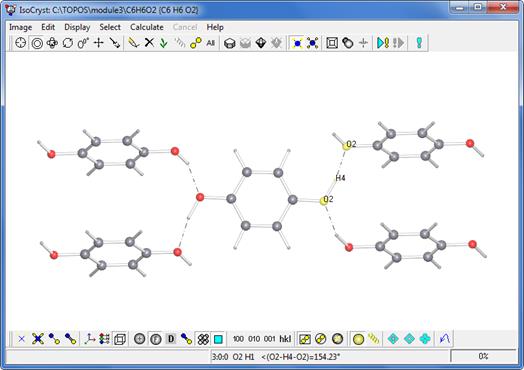
 и выделяйте пары, тройки или четвёрки атомов. Значение параметра выводится в статус-строке. Например, ниже приведено значение угла для водородной связи O2–H4¼O2 равное 154.23° (посмотрите на статус-строку внизу окна).
и выделяйте пары, тройки или четвёрки атомов. Значение параметра выводится в статус-строке. Например, ниже приведено значение угла для водородной связи O2–H4¼O2 равное 154.23° (посмотрите на статус-строку внизу окна).

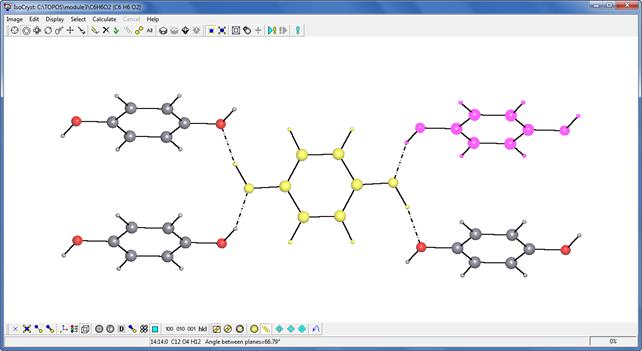
Для вычисления двугранного угла между двумя плоскостями выделите атомы, находящиеся в одной плоскости (выделены желтым цветом), затем выделите атомы другой плоскости, удерживая клавишу Ctrl (выделены малиновым цветом). Таким образом, двугранный угол между двумя молекулами гидрохинона равен 66.79°. Для удаления малинового выделения, нажав клавишу Ctrl, щелкните правой кнопкой мыши на атомах или на свободном месте рабочего окна.

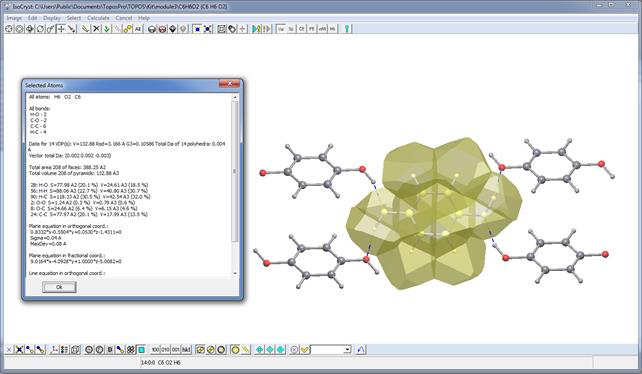
- Выделите молекулу гидрохинона желтым цветом и нажмите кнопку
 . Будет создан молекулярный полиэдр Вороного-Дирихле. Откройте карточку Information, выбрав Calculate/Selected Atoms,и найдите в полученной информации объем молекулы (V = 131.99 или 132.88 Å3 для двух неэквивалентных молекул). Нажмите Ok длязакрытия окна.
. Будет создан молекулярный полиэдр Вороного-Дирихле. Откройте карточку Information, выбрав Calculate/Selected Atoms,и найдите в полученной информации объем молекулы (V = 131.99 или 132.88 Å3 для двух неэквивалентных молекул). Нажмите Ok длязакрытия окна.
Теперь отмените выделение молекулы, чтобы получить следующее изображение.
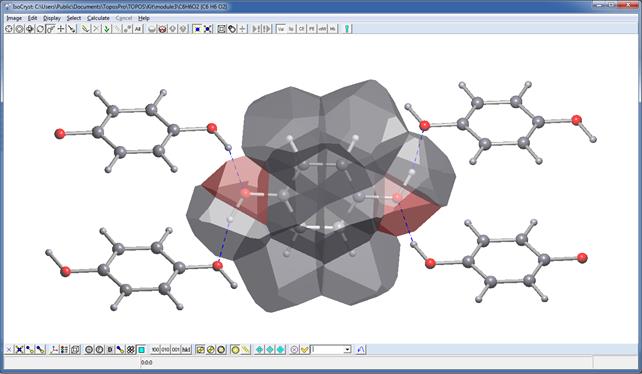
Удалите изображение полиэдра Вороного-Дирихле нажатием кнопки  .
.
(4) Закройте окно программы IsoCryst.
Упражнение: проанализируйте Ваши собственные структуры с помощью программы IsoCryst.
Рекомендации: Ознакомьтесь с назначением других кнопок, расположенных на нижней панели:
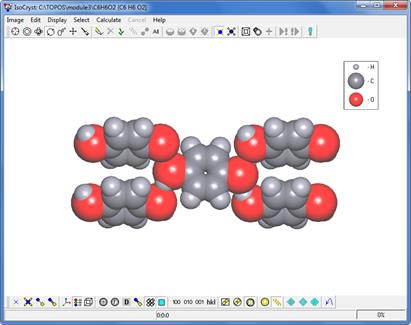
 Increase Atom radius. (Увеличение атомного радиуса.) В разделе Radii System установите флаг Sph. Domain, чтобы получить размер атома близкий к заполнению пространства. При небольшом изменении параметров Вы можете получить изображение CPK такое, как показано ниже.
Increase Atom radius. (Увеличение атомного радиуса.) В разделе Radii System установите флаг Sph. Domain, чтобы получить размер атома близкий к заполнению пространства. При небольшом изменении параметров Вы можете получить изображение CPK такое, как показано ниже.
 Show Vertices (Показывать Вершины) и Fill Faces (Заливка Граней), начинают действовать на полиэдре Вороного-Дирихле.
Show Vertices (Показывать Вершины) и Fill Faces (Заливка Граней), начинают действовать на полиэдре Вороного-Дирихле.
|
|
|
© helpiks.su При использовании или копировании материалов прямая ссылка на сайт обязательна.
|